細胞内分子動態の可視化を利用した細胞間情報共有システムの解明
研究代表者(所属):溝口貴正(千葉大学 薬学研究院 生化学研究室・助教)
研究内容

1.細胞骨格ダイナミクス、細胞内シグナル活性のイメージング
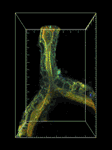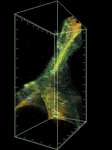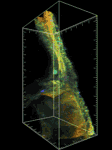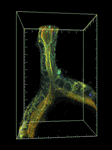
細胞内分子の動態や細胞内シグナルの活性化を可視化できる既存の蛍光プローブを利用して、細胞骨格、細胞内シグナル活性をライブイメージング可能なトランスジェニックラインを整備する。次に移動中のゼブラフィッシュ側線原基におけるプローブの細胞内分布や活性化のパターンを経時的にメージングし、例えば「アクチンフィラメントは移動方向に強く集積する」、「Rasの活性化は隣り合う細胞に伝達していく」といった一定のパターン秩序が見られるか観察する。
2.細胞情報共有システムを担う分子メカニズムの解明
遺伝子の機能阻害や化学物質によるシグナル阻害実験を行い、移動中のゼブラフィッシュ側線原基内におけるパターン秩序が崩壊するか検討する。
以上の解析より、本研究では細胞間での情報共有に関与する分子メカニズムの解明を目指す。
代表的論文3編
Neuron and sensory epithelial cell fate is sequentially determined by Notch signaling in zebrafish lateral line development.
Mizoguchi, T., Togawa, S., Kawakami, K. and Itoh, M.
J Neurosci, 31, 15522-15530, (2011)
Mib-Jag1-Notch signalling regulates patterning and structural roles of the notochord by controlling cell-fate decisions.
Yamamoto, M., Morita, R., Mizoguchi, T., Matsuo, H., Isoda, M., Ishitani, T., Chitnis, A.B., Matsumoto, K., Crump, J.G., Hozumi, K., Yonemura, S., Kawakami, K. and Itoh, M.
Development, 137, 2527-2537, (2010)
Sdf1/Cxcr4 signaling controls the dorsal migration of endodermal cells during zebrafish gastrulation.
Mizoguchi, T., Verkade, H., Heath, J., Kuroiwa, A. and Kikuchi, Y.
Development, 135, 2521-2529, (2008)
関連HP 千葉大学 薬学研究院 生化学研究室(http://www.p.chiba-u.ac.jp/lab/seika/index.html)